栄養ストレスに応じた植物細胞内リン酸化シグナルダイナミクスを網羅的に解明
形態機能学系の佐藤長緒先生と山口淳二先生らの研究グループは、植物の栄養ストレス応答に関する新しい知見を論文発表しました。詳しい日本語解説はこちらから読むこともできますが、以下、佐藤先生による解説です。
私達は、国内および海外の研究者らとの協力しながら、生命を支える複雑な細胞内システムを包括的に理解する取組みを始めています。
本研究では、細胞内のリン酸化シグナルダイナミクスを網羅的に解析し、重要な代謝物の変動や遺伝子発現解析と結びつけることで、これまで未知であった植物細胞内の栄養応答シグナル伝達ネットワークの全体像を理解することに成功しました。
具体的には、生物に必須の栄養素である糖(炭素、C)と窒素(N)のバランス「C/Nバランス」に着目して、この栄養バランスの乱れにより起こるストレスに応答した細胞内リン酸化ダイナミクスを網羅的に解析しました。
真核生物の細胞内において、タンパク質のリン酸化修飾は、細胞内シグナル伝達系の分子スイッチとして重要な役割を果たします。実際にこれまでの私達の研究からも、植物の栄養シグナル伝達に関与する鍵因子としてリン酸化酵素(キナーゼ)やその標的タンパク質が発見されています。その一方で、数千を超える細胞内のタンパク質を対象としてグローバルなリン酸化シグナル変動の実態を把握することは、これまでの個別遺伝子の機能解析だけでは困難であり、未知のものでした。
本研究では、先端的なマススペクトロメトリー装置を用いたリン酸化プロテオーム解析という手法を活用することで、細胞内のリン酸化シグナルの網羅的な検出と定量的な比較解析を行いました。その結果、1785のリン酸化ペプチドを検出し、193個のタンパク質のリン酸化レベルがC/Nバランスに応じて変化していることが分かりました。その中には、糖や窒素代謝制御に関わる代謝酵素群や輸送体、タンパク質合成(翻訳)関連因子が含まれており、加えて、転写因子やキナーゼといった多くのシグナル伝達系タンパク質が同定されました(図1)。
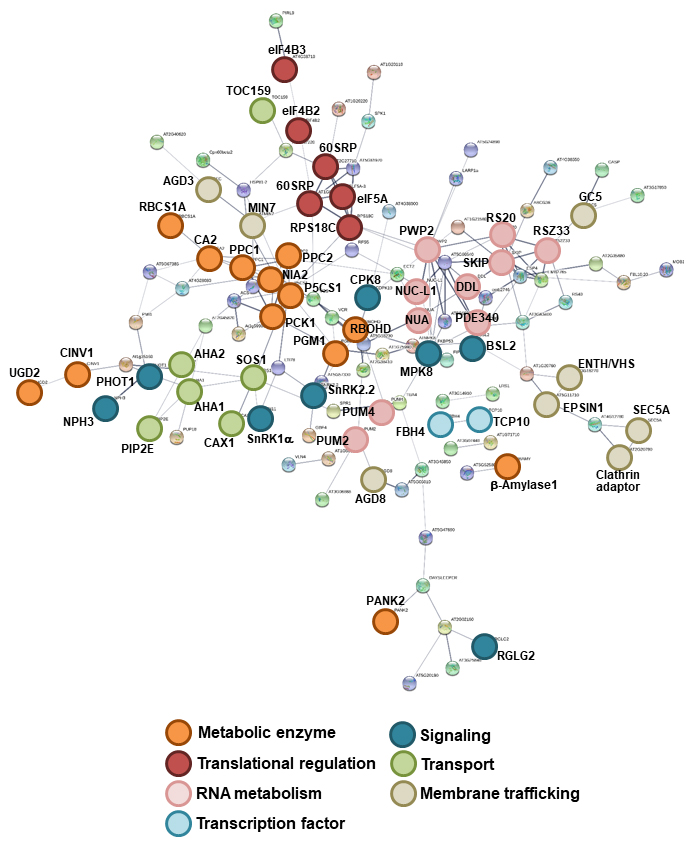 図1 C/N応答性リン酸化タンパク質ネットワーク
図1 C/N応答性リン酸化タンパク質ネットワーク
こうしたリン酸化プロテオーム解析の結果から、C/Nシグナルの上流制御因子としてSnRK1キナーゼ(酵母Snf1、哺乳類AMPKホモログ)が重要な役割を果たすことを見出しました。
また、新規の受容体型キナーゼLMK1(Leucine-rich repeat Malectin Kinase 1と命名)を単離し、このキナーゼが細胞死制御に関与することを発見しました。細胞死は、植物の病原体抵抗性や老化制御といった生理現象で非常に重要な意義を有します。今回単離したLMK1は、受容体型キナーゼとして、栄養シグナルとこうした生理現象を結びつける鍵因子として働いている可能性が示唆されます。
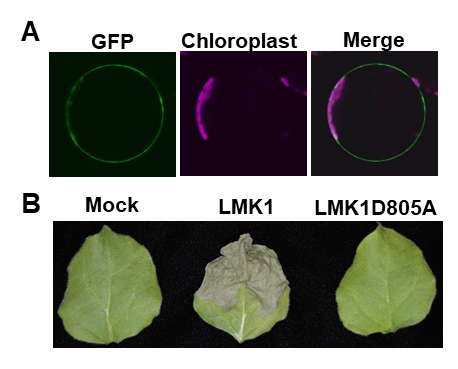 図2 新規受容体型キナーゼLMK1に細胞死誘導
図2 新規受容体型キナーゼLMK1に細胞死誘導
(A)LMK1-GFPタンパク質細胞内局在性の蛍光顕微鏡観察
(B)LMK1過剰発現による細胞死誘導(LMK1D805A:キナーゼドメイン変異型LMK1)
現在このリン酸化プロテオーム解析で得られたC/Nシグナル関連因子群の機能に関して、さらに深く研究を進めています。
今回取得したリン酸化プロテオーム解析データは、植物栄養応答の分子機構を理解するためのプラットフォーム構築に貢献し、他の作物種も含めた様々な応用研究の基盤情報となることが期待されます。
なお本研究は、北海道大学、理化学研究所、Max-Planck-Institute for Plant Breeding Research(ドイツ)、Max Planck Institute of Molecular Plant Physiology(ドイツ)、奈良先端科学技術大学院大学、Hohenheim大学(ドイツ)との共同研究として実施されました。
論文:Li X, Sanagi M, Lu Y, Nomura Y, Stolze SC, Yasuda S, Saijo Y, Schulze WX, Regina F, Stitt M, Lunn JE, Nakagami H*, Sato T*, Yamaguchi J (2020) Protein phosphorylation dynamics under carbon/nitrogen-nutrient stress and identification of a cell death-related receptor-like kinase in Arabidopsis. Frontiers in Plant Science 11: 377. (*Corresponding authors) doi: 10.3389/fpls.2020.00377.
